Corona-Infektionsverlauf im Ländervergleich
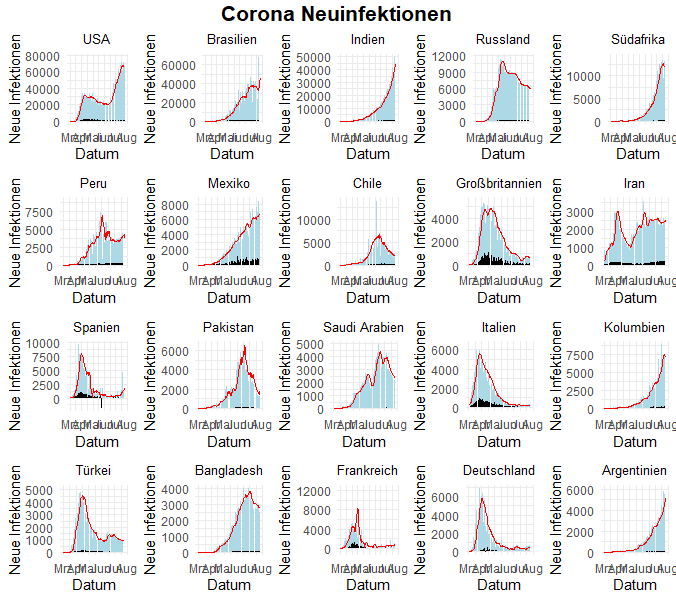
Die Corona-Pandemie verläuft in den Ländern ganz unterschiedlich. Grafiken der 20 Länder mit den meisten Infektionen (Stand 1. August 2020) zeigen gut die unterschiedlichen Verläufe.
Die Daten stammen unter anderem von der Johns Hopkins University in Baltimore. Das Package tidycovid19 liest die Daten ein. Es ist auf Github erhältlich. Mit Hilfe von gridExtra werden die 20 einzelnen Diagramme zu einer großen Grafik zusammengefasst.
library(tidyverse)
library(tidycovid19)
library(zoo)
library(grid)
library(gridExtra)
df <- download_merged_data(cached = TRUE, silent = TRUE)
p1 <- df %>%
filter(iso3c == "USA") %>%
mutate(
new_cases = confirmed - lag(confirmed),
new_deaths = deaths - lag(deaths),
ave_new_cases = rollmean(new_cases, 7, na.pad=TRUE, align="right")) %>%
filter(!is.na(new_cases), !is.na(ave_new_cases), !is.na(new_deaths), date > "2020-03-01") %>%
ggplot(aes(x = date)) +
geom_bar(aes(y = new_cases), stat = "identity", fill = "lightblue") +
geom_bar(aes(y = new_deaths), stat = "identity", fill = "black") +
geom_line(aes(y = ave_new_cases), color ="red") +
theme_minimal() +
ggtitle("USA") +
theme(plot.title = element_text(hjust = 0.5, size = 10)) +
xlab("Datum") +
ylab("Neue Infektionen")
p2 <- df %>%
filter(iso3c == "BRA") %>%
mutate(
new_cases = confirmed - lag(confirmed),
new_deaths = deaths - lag(deaths),
ave_new_cases = rollmean(new_cases, 7, na.pad=TRUE, align="right")) %>%
filter(!is.na(new_cases), !is.na(ave_new_cases), !is.na(new_deaths), date > "2020-03-01") %>%
ggplot(aes(x = date)) +
geom_bar(aes(y = new_cases), stat = "identity", fill = "lightblue") +
geom_bar(aes(y = new_deaths), stat = "identity", fill = "black") +
geom_line(aes(y = ave_new_cases), color ="red") +
theme_minimal() +
ggtitle("Brasilien") +
theme(plot.title = element_text(hjust = 0.5, size = 10)) +
xlab("Datum") +
ylab("Neue Infektionen")
p3 <- df %>%
filter(iso3c == "IND") %>%
mutate(
new_cases = confirmed - lag(confirmed),
new_deaths = deaths - lag(deaths),
ave_new_cases = rollmean(new_cases, 7, na.pad=TRUE, align="right")) %>%
filter(!is.na(new_cases), !is.na(ave_new_cases), !is.na(new_deaths), date > "2020-03-01") %>%
ggplot(aes(x = date)) +
geom_bar(aes(y = new_cases), stat = "identity", fill = "lightblue") +
geom_bar(aes(y = new_deaths), stat = "identity", fill = "black") +
geom_line(aes(y = ave_new_cases), color ="red") +
theme_minimal() +
ggtitle("Indien") +
theme(plot.title = element_text(hjust = 0.5, size = 10)) +
xlab("Datum") +
ylab("Neue Infektionen")
...
...
...
p20 <- df %>%
filter(iso3c == "ARG") %>%
mutate(
new_cases = confirmed - lag(confirmed),
new_deaths = deaths - lag(deaths),
ave_new_cases = rollmean(new_cases, 7, na.pad=TRUE, align="right")) %>%
filter(!is.na(new_cases), !is.na(ave_new_cases), !is.na(new_deaths), date > "2020-03-01") %>%
ggplot(aes(x = date)) +
geom_bar(aes(y = new_cases), stat = "identity", fill = "lightblue") +
geom_bar(aes(y = new_deaths), stat = "identity", fill = "black") +
geom_line(aes(y = ave_new_cases), color ="red") +
theme_minimal() +
ggtitle("Argentinien") +
theme(plot.title = element_text(hjust = 0.5, size = 10)) +
xlab("Datum") +
ylab("Neue Infektionen")
grid.arrange(p1, p2, p3, p4, p5, p6, p7, p8, p9, p10,
p11, p12, p13, p14, p15,
p16, p17, p18, p19, p20, ncol=5, top=textGrob("Corona Neuinfektionen",
gp=gpar(fontsize=16, fontface = "bold")))
Das Skript: Das Package tidyverse ist ein Meta-Package, das mehrere Packages wie ggplot2 und dplyr nachlädt. Die Installation von tidycovid19 kann über devtools::install_github("joachim-gassen/tidycovid19") erfolgen. download_merged_data() liefert einen Länder-Tages-Data-Frame, der Daten aus verschiedenen Quellen zusammenführt. Der Data Frame wird entsprechend angepasst. Das darzustellende Land wird als ISO3c-Code übergeben. Anschließend erfolgt die Berechnung der täglichen Neuinfektionen sowie der Todeszahlen. Das Startdatum ist hier der 1. März 2020. Anschließend erzeugt ggplot das Diagramm. Der Block wird 19 Mal wiederholt, wobei lediglich der ISO3c-Code sowie der Titel des Diagramms zu ändern sind. Schließlich erstellt grid.arrange() aus den 20 Einzeldiagrammen die endgültige Grafik. Die Diagramme sind in 4 Reihen à 5 Diagramme angeordnet. Das lässt sich gut als Querformat ausdrucken.
 Schreiben macht durstig! Spendier mir doch ein Bier, indem du mir einen kleinen Betrag via Paypal schickst oder ein paar Bitcoin- oder Ethereum-Bruchteile an die entsprechende nachfolgende Adresse. Prost & auf dein Wohl!
Schreiben macht durstig! Spendier mir doch ein Bier, indem du mir einen kleinen Betrag via Paypal schickst oder ein paar Bitcoin- oder Ethereum-Bruchteile an die entsprechende nachfolgende Adresse. Prost & auf dein Wohl!
Bitcoin-Adresse : 1PjYNNjHmkAUmf626hukL32Vs1wFt8v1wT
Ethereum-Adresse : 0x974e73994c7483eef1396a21b9937aecb7b6c286
Schreibe einen Kommentar